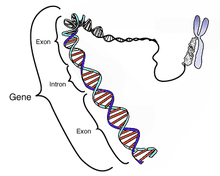
Un intron est une portion d'un gène qui est transcrite en ARN, au sein d'un ARN précurseur, et qui est ensuite éliminée par un processus d'excision programmé et qu'on ne retrouve donc pas dans l'ARN mature. On trouve principalement des introns dans les gènes codant des protéines, où ils sont présents dans l'ARN prémessager et excisés dans l'ARNm mature[1]. Les introns sont donc des régions non codantes. On trouve aussi des introns dans des gènes codant des ARN non codants comme les ARN ribosomiques ou les ARN de transfert.
Un gène pourvu d'introns est appelé gène discontinu, gène fragmenté ou gène mosaïque.
Le processus d'excision des introns s'appelle l'épissage. Les segments d'ARN qui sont conservés après épissage des introns sont appelés des exons.
On trouve principalement des introns dans les gènes codant des protéines des organismes eucaryotes. Les gènes eucaryotes sont constitués d'une alternance d'exons et d'introns, commençant et terminant par un exon.
- Exon 1
- Intron 1
- Exon 2
- Intron 2
- …
- Intron n-1
- Exon n.
Après la transcription, l'ARN précurseur synthétisé va subir un certain nombre de modifications, dont l'épissage, au cours duquel les introns vont être excisés de l'ARN. Les exons vont quant à eux être suturés pour donner l'ARN mature par ce mécanisme d'épissage. On obtiendra donc un ARN de type Exon 1 - Exon 2 - Exon 3 - ... - Exon n.

Les introns ne jouent donc aucun rôle dans la fonction de l'ARN mature (traduction en protéine pour l'ARNm, incorporation dans le ribosome pour les ARNr...), si bien que leur fonction éventuelle reste à ce jour difficile à cerner. Leur rôle le plus important est de permettre une combinatoire lors de l'épissage. Cela permet à certains gènes de coder plusieurs protéines ou variants d'une même protéine, par épissage alternatif d'un même ARN pré-messager. Ceci permet par exemple à certains rétrovirus de produire plusieurs ARNm et donc plusieurs protéines virales à partir d'un seul promoteur de transcription et donc d'un seul pré-ARNm.
La taille des introns est très variable, allant de quelques dizaines de paires de bases, jusqu'à plusieurs dizaines de milliers[2]. La taille moyenne varie suivant les espèces et a tendance à augmenter avec la taille du génome[2].
De manière plus rare, on retrouve également des introns dans certains gènes chez les archéobactéries et chez les procaryotes[3]. Il s'agit d'introns d'un type particulier, dits introns autoépissables.
- (en) Bruce Alberts, Molecular Biology of the cell : Sixth International Student Edition, Garland Science, , 1465 p. (ISBN 978-0-8153-4464-3, lire en ligne), Chapter 6 : How cells read the genome : From DNA to protein, "RNA splicing is performed by the Spliceosome", p.319
- (en) Xin Hong, Douglas G. Scofield et Michael Lynch, « Intron size, abundance, and distribution within untranslated regions of genes », Molecular Biology and Evolution, vol. 23, no 12, , p. 2392–2404 (ISSN 0737-4038, PMID 16980575, DOI 10.1093/molbev/msl111, lire en ligne, consulté le )
- F. Martínez-Abarca et N. Toro, « Group II introns in the bacterial world », Molecular Microbiology, vol. 38, no 5, , p. 917–926 (ISSN 0950-382X, PMID 11123668, lire en ligne, consulté le )